| Interface Gráfica, DICEWin | |
|
A interface gráfica foi desenvolvida com o objetivo de facilitar
o utilização dos programas DICE e ORDER, e o manuseio dos
arquivos de entrada e saída das simulações.
DICEWin
é
compatível com plataforma WINDOWS e LINUX através do ambiente
de desenvolvimento JAVA. Desta forma, para a interface funcionar é
necessária a instalação do
JAVA
.
DiceWin possui 4 módulos:
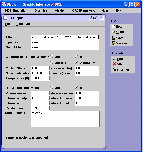
Esse módulo também chama a execução externa do DICE, que gera vários arquivos de saída com dados em formatos diferentes que serão graficados nos módulo Graphic e no módulo Vismol. Adicionalmente, o principal arquivo de saída (*.out) é mostrado em tempo real. O módulo Graphic lê os arquivos gerados pelo DICE e pelo programa ORDER que realiza a análise das configurações moleculares posteriormente a simulação e mostra na tela os resultados graficamente. Os arquivos podem ter 5 formatos diferentes que são identificados pela terminação dos arquivos: *.out, *.gr, *.dst, *.hbd e outros. Cada um desses arquivos com formatos diferentes podem ter mais de um conjunto de dados a ser graficado individualmente ou em até conjunto de três. Esses gráficos são mostrados em forma de dispersão, com símbolos e linhas, e histograma. Veja um exemplo na figura abaixo. Nesse módulo, também existe uma opção para a atualização automática dos gráficos que permitem ao usuário ver a evolução da simulação em tempo real. Outras opções interessantes são: (i) selecionar o arquivo de leitura através de janela com estrutura de diretórios, como é padrão nos ambientes gráficos de sistema operacional como Windows, KDE, etc; (ii) salvar separadamente um conjunto de dado em um arquivo; (iii) ter as coordenadas dos pontos através do click do mouse no gráfico (no caso particular do gráfico da função de distribuição radial de pares, G(r), também é possível ter o valor da integração do pico através da seleção do mouse na curva). No módulo Vismol foram feitas várias modificações no algoritmo, que o tornaram mais eficiente no sentido de que o algoritmo é capaz de ler várias configurações moleculares dentro de um único arquivo, mostra o número de configurações moleculares, permite ao usuário escolher qual configuração deseja visualizar ou se deseja ver o filme com a animação de todas as configurações lidas. Para que o usuário realize essas tarefas, foi introduzido uma barra de ferramentas com os botões referentes a suas determinadas funções. Veja ilustração na figura abaixo. Nesse módulo, também existem várias opções de visualização que permitem: (i) mudar cor de fundo e cor de átomos selecionados; (ii) mudar o modelo das moléculas de átomos (círculos) e ligações (retângulos), para só átomos, só ligações ou só linhas; (iii) rotacionar em qualquer direção, transladar e re-escalar, etc. O módulo Order é utilizado para a inserção de dados para análise das configurações moleculares pelo programa ORDER (desenvolvido para auxiliar o DICE). Veja ilustração desse módulo na figura abaixo. Esse módulo também chama a execução externa do ORDER, que: (i) separa configuração estatisticamente descorrelacionadas, (ii) para cada configuração separa camadas de solvatação esféricas ou ajustadas ao formado do soluto, (iii) identifica e separa moléculas que fazem ligação de Hidrogênio com o soluto, (iv) salva arquivos em diferentes formatos de leitura de outros programa como GAUSSIAN, ZINDO e MOLCAS. Esse módulo é uma ferramenta essencial de análise após as simulações. É interessante ressaltar que com essa interface é possível utilizar o DICE não só com fins científicos mas também com fins pedagógico. É possível realizar “Experimentos Computacionais” através de algumas simulações de líquidos moleculares para alunos de graduação de cursos de Química e Física. |
|
|
|
|
 Click para ampliar
Click para ampliar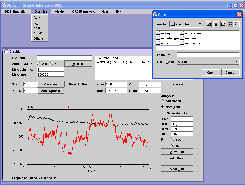 Click para ampliar
Click para ampliar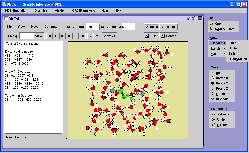 Click para ampliar
Click para ampliar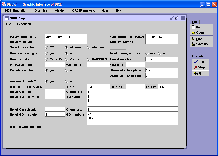 Click para ampliar
Click para ampliar